Durée de l’activité : 30 minutes
Âge conseillé : dès 15 ans
Population geneticsMedical Data Science
Chasse aux variants
- A vous de jouer
Un nouveau variant est là ! Trouvez les différences, leur impact et retracez l'évolution du coronavirus...
Activité 1
Cherchez les différences !
Chaque fois que SARS-CoV-2 infecte une cellule humaine, des milliers de copies de lui-même, et donc de son matériel génétique, sont produites…et ces copies ne sont pas toujours identiques !
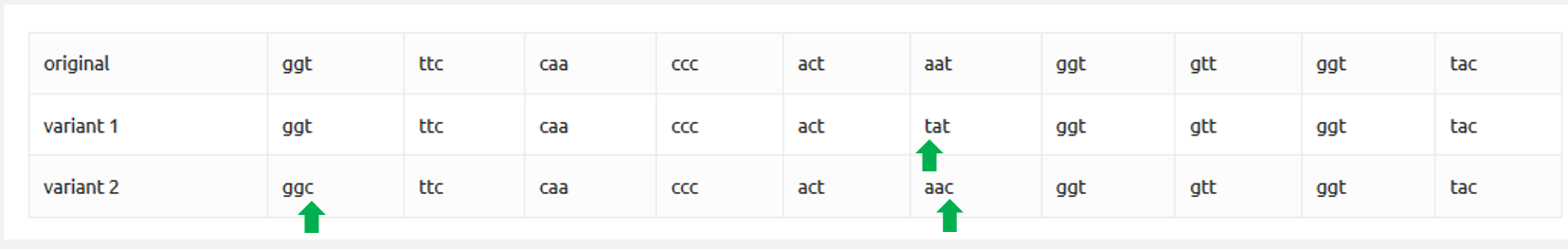
Voici 3 petits morceaux du matériel génétique de SARS-CoV-2 :
l’original et celui retrouvé dans deux copies différentes (variant 1 et variant 2), provenant de 2 villes différentes.
| original | ggt | ttc | caa | ccc | act | aat | ggt | gtt | ggt | tac |
| variant 1 | ggt | ttc | caa | ccc | act | tat | ggt | gtt | ggt | tac |
| variant 2 | ggc | ttc | caa | ccc | act | aac | ggt | gtt | ggt | tac |
Question 1
Combien de différences?
Réponse
Il y a 3 différences (en vert):
Pour les plus motivés…
Voici l’alignement du génome de SARS-CoV-2 ‘original’ et du génome du variant Alpha :
amusez-vous à compter les différences entre ces 2 séquences de 29’903 nucléotides…
Truc: la présence d’un * signifie qu’il n’y a pas de différence, la présence d’un – signifie qu’il manque une lettre.
Activité 2
Une différence peut faire toute la différence !
Le matériel génétique du virus contient des recettes pour fabriquer des protéines. Les différences (appelées mutations) retrouvées dans le matériel génétique des virus variants peuvent modifier les protéines produites à partir de ces recettes…mais pas toujours !
Une protéine présente à la surface du virus, la protéine spike, est particulièrement surveillée par les scientifiques. C’est elle qui permet au virus de pénétrer dans les cellules humaines et c’est elle qui est également reconnue par les anticorps que nous produisons pour lutter contre le virus !
Selon les différences, le virus pourrait devenir plus contagieux ou même échapper aux vaccins !
Traduire les 3 séquences de nucléotides en séquence d’acides aminés en utilisant le code génétique :
compléter les acides aminés manquants dans la séquence de la protéine spike.
Le code génétique
3 lettres nucléotides (A,T,C,G) correspondent à une lettre acide aminé (G,E,N,I,A, L, ...)
Pour traduire, partez du milieu vers l’extérieur.
Exemple : GTT -> V
| position | 496 | 497 | 498 | 499 | 500 | 501 | 502 | 503 | 504 | 505 |
| nucléotides (original) | ggt | ttc | caa | ccc | act | aat | ggt | gtt | ggt | tac |
| acides aminés (original) | G | F | Q | P | T | N | G | V | G | Y |
| nucléotides (variant 1) | ggt | ttc | caa | ccc | act | tat | ggt | gtt | ggt | tac |
| acides aminés (variant 1) | G | G | V | G | Y | |||||
| nucléotides (variant 2) | ggc | ttc | caa | ccc | act | aac | ggt | gtt | ggt | tac |
| acides aminés (variant 2) | P | G | V | Y |
Question 2
Combien de différences entre les séquences de protéine ?
Faites le lien avec le nombre de différences de la Question 1...
Réponse
Il y a 3 différences entre les séquences de nucléotides (en vert) mais une seule différence entre les séquences en acides aminés : N -> Y (en rouge).
Une mutation dans une séquence de nucléotides ne modifie donc pas forcément la séquence en acides aminés de la protéine correspondante, car le code génétique est ‘redondant’. Heureusement !
| …496 | 497 | 498 | 499 | 500 | 501 | 502 | 503 | 504 | 505… | |
| nucléotides (original) | ggt | ttc | caa | ccc | act | aat | ggt | gtt | ggt | tac |
| acides aminés (original) | G | F | Q | P | T | N | G | V | G | Y |
| nucléotides (variant 1) | ggt | ttc | caa | ccc | act | tat | ggt | gtt | ggt | tac |
| acides aminés (variant 1) | G | F | Q | P | T | Y | G | V | G | Y |
| nucléotides (variant 2) | ggc | ttc | caa | ccc | act | aac | ggt | gtt | ggt | tac |
| acides aminés (variant 2) | G | F | Q | P | T | N | G | V | G | Y |
La mutation N->Y retrouvée dans le virus ‘variant 1’ se trouve en position 501 de la séquence en acides aminés de la protéine spike : elle est appelée N501Y.
Cette mutation pourrait avoir un impact sur la capacité de SARS-CoV-2 à infecter les cellules humaines, car elle est située dans la région de la protéine spike qui lie les cellules humaines.
Activité 3
Séquencer le matériel génétique et trouver les mutations permet aussi de reconstruire l’histoire de la transmission du virus et donc de suivre son évolution !
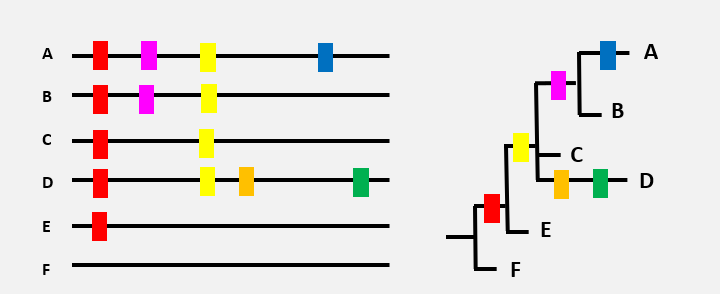
Voici une représentation très shématique du matériel génétiques de SARS-CoV-2, chacun avec une combinaison différente de mutations (rectangles de différentes couleurs). Le variant F est ‘l’original’.
L’ordre d’apparition des différentes mutations peut être représenté par un arbre. La mutation rouge, présente chez tous les virus variants est probablement apparue en premier (variant E), puis la mutation jaune (variant C),…
Question 3
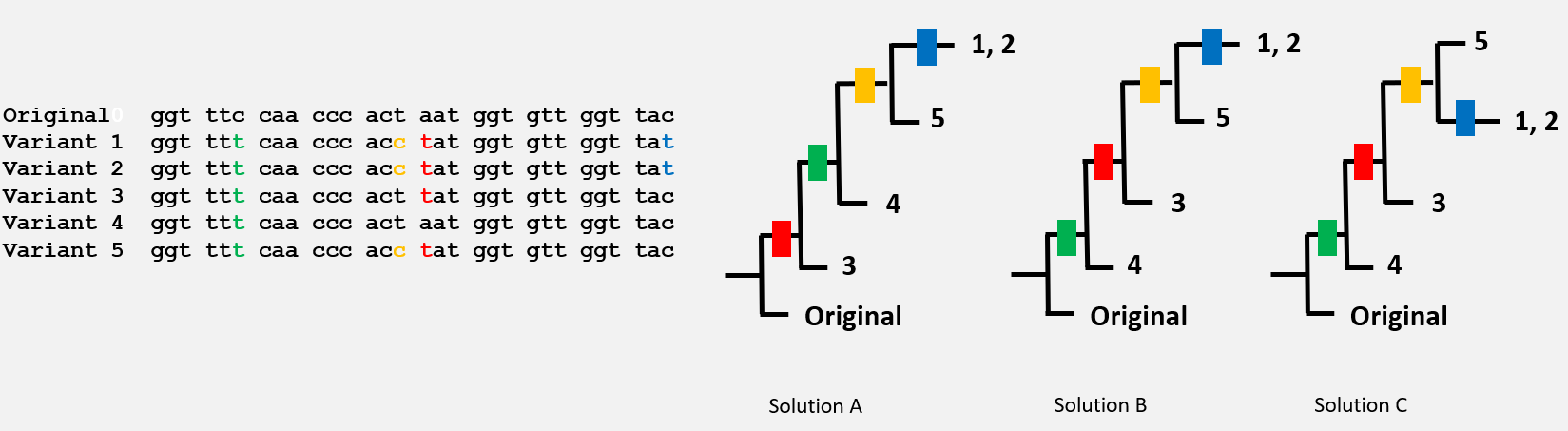
Quelle est la bonne solution ?
Voici les séquences de nucléotides de 5 virus variants. Quel est l'arbre d'apparation des mutations qui vous semble le plus juste ?
Réponse
La réponse correcte est ‘Solution B’ et ‘Solution C’ : ces 2 arbres sont identiques : la branche du haut, un peu comme un mobile a été inversée !
Le variant 4 est probablement apparu avant les autres !
Activité 4
Voici une séquence de SARS-CoV-2 retrouvée dans les eaux usées d’une station d’épuration d’une station de ski suisse début décembre 2020 :
| position | 496 | … | … | … | … | … | … | … | … | 505 |
| codon | ggt | ttc | caa | ccc | act | tat | ggt | gtt | ggt | tac |
La mutation N501Y est-elle présente ? Pourrait-il s’agir du variant Alpha ?
Truc : utilisez le code génétique pour traduire la séquence…
Réponse
Protéine spike : voici la position des différents codons et et des acides aminés correspondants :
| position | 496 | 497 | 498 | 499 | 500 | 501 | 502 | 503 | 504 | 505 |
| codon | ggt | ttc | caa | ccc | act | tat | ggt | gtt | ggt | tac |
| acide aminé | G | F | Q | P | T | Y | G | V | G | Y |
La séquence de la protéine contient l’acide aminé Y en position 501, ce qui correspond à la fameuse mutation N501Y.
Pour la petite histoire, cette mutation (et plusieurs autres mutations présentes dans le variant Alpha) a été retrouvée dans des échantillons d’eaux usées provenant de Lausanne et d’une station de ski suisse. Ces échantillons avaient été collectés entre juillet et décembre 2020. Ces résultats suggèrent que le variant Alpha pourrait déjà avoir circulé en Suisse fin 2020. Or, le variant Alpha a été officiellement découvert en Angleterre début décembre 2020 !
