Dernière mise à jour: avril 2022
Paleogenomics
Dinopoulet - Histoire
Les dinosaures n’auraient pas tous disparu ? Vraiment ?
Contexte
Une extinction de masse a eu lieu il y a environ 66 millions d’années, au printemps, causée par l’impact de l’astéroïde ‘Chicxulub impactor’ au Yucatan (Mexique).
Cet évènement a provoqué la disparition d’environ 75 % des espèces, dont les mollusques marins appelés ammonites et la plupart des dinosaures et des reptiles marins.
Mais les dinosaures n’ont pas tous disparu : certains ont survécu. Que sont devenus leurs descendants ?
Voici quelques éléments de réponse, basés sur des petits bouts de séquences de protéines retrouvées dans des os fossilisés de Brachylophosaurus canadensis et de Tyrannosaurus rex, deux espèces de dinosaures qui auraient disparu il y a respectivement quelque 80 et 68 millions d’années !
Podcast : C’est au printemps que les dinosaures ont été rayés de la Terre (RTS, mars 2022) – Source : The Mesozoic terminated in boreal spring (2022)
Une collection de fossiles
Les scientifiques collectent des informations sur les dinosaures et les espèces aujourd’hui disparues grâce aux différents fossiles retrouvés un peu partout sur notre planète : ils récupèrent ainsi des données morphologiques et parfois même des données moléculaires (ADN ou protéine), qu’ils peuvent comparer afin de déterminer les liens de parenté.
C'est quoi un fossile ?
Les fossiles sont des traces plus ou moins minéralisées d’anciens organismes vivants : cela peut être des os, des dents, des œufs, des traces de leur activité passée (emprunte, coprolithe, …) ou des moulages, conservés dans une roche sédimentaire.
Et certains de ces fossiles contiennent encore de l’ADN ou des protéines !
Quelques dates clés
Voici un petit récapitulatif pour mieux s’y retrouver dans l’histoire de la vie, des dinosaures … et des plus vieux fossiles :
Des protéines ‘fossiles’ ?
Il est aujourd’hui possible de séquencer les protéines retrouvées dans des fossiles de coquille d’œuf, de dent, d’os, ou de tissus momifiés : c’est le domaine appelé en anglais ‘Deep Time Paleoproteomics’…Et les records commencent à s’accumuler car les protéines semblent mieux se conserver que l’ADN au fil du temps et les technologies sont de plus en plus performantes !
Source: Deep Time Paleoproteomics: Looking Forward (2022)
Séquencer une protéine ?
A l’image d’un collier de perle, une protéine est constituée d’une succession d’acides aminés liés les uns aux autres par des liaisons chimiques. Il existe 20 acides aminés différents, représentés par des lettres (G, E, N, I , A, L, …).
La spectométrie de masse est une technologie qui permet de séquencer les protéines, c’est-à-dire de connaître l’ordre dans lequel s’enchaînent les acides aminés.
Les protéines célèbres en paléontologie...
Les protéines étudiées par les scientifiques qui s’intéressent aux organismes disparus sont en particulier le collagène et le cytochrome.
Pourquoi ?
Le collagène est une des protéines les plus abondantes dans les os. C’est lui qui est le plus fréquemment retrouvé dans la matière organique présente dans les os fossilisés.
Voici la protéine collagène de différentes espèces et la protéine collagène d’espèces aujourd’hui disparues dans la banque de données UniProtKB.
—
L’information que l’on a sur certaines protéines provient également de l’ADN. Si on connaît une séquence ADN, on peut prédire la séquence de la protéine correspondante. L’ADN est retrouvé surtout dans le noyau des cellules eucaryotes, mais également dans des organelles comme les mitochondries ou les chloroplastes. Or l’ADN retrouvé dans les mitochondries se conserve mieux que l’ADN présent dans le noyau des cellules au cours du temps. La protéine cytochrome est une des protéines dont le gène se trouve dans l’ADN mitochondrial.
Voici par exemple l’ADN mitochondrial de l’homme de Neandertal et la séquence du cytochrome de l’homme de Neandertal ou encore, la séquence des cytochromes du Dodo et du loup de Tasmanie.
Voici la liste des protéines provenant d’organismes aujourd’hui disparus dans la banque de données UniProtKB.
Voici des fragments de la protéine collagène retrouvée chez différentes espèces.
Tyrannosaurus rex
GATGAPGIAGAPGFPGARGAPGPQGPSGAPGPKGSAGPPGATGFPGAAGRGVQGPPGPQGPR
Datation: 68 millions d’années
Gallus gallus (poulet)

GATGAPGIAGAPGFPGARGPSGPQGPSGAPGPKGNSGEPGAPGNKGDTGAKGEPGPAGVQGP
Datation: 10’000 à 50’000 ans
Mammut americanum (Mammouth)

GANGAPGIAGAPGFPGARGPAGPQGPSGAPGPKGNSGEPGAPGSKGDAGAGIQGPPGPAGEE
Datation: 1,65 million d’années
Brachylophosaurus canadensis
GATGAPGIAGAPGFPGARGPSGPQGPSGAPGPKGSAGPPGATGFPGAAGRGETGPAGPAGPP
Datation: 80 millions d’années
L’évolution du collagène
Pour définir les liens de parenté, les biologistes recherchent ce que les espèces ont en commun mais aussi ce qui les distingue.
La protéine collagène est présente entre autre dans les os de tous les vertébrés. Mais comme toutes les protéines, elle est un peu différente d’une espèce à l’autre. Ces différences sont une véritable mine d’or pour étudier l’évolution et les liens de parenté qui existent entre les différentes espèces.
Comment étudier les liens de parenté entre espèces?
On peut, par exemple, compter les différences entre les séquences de collagène provenant de ces différentes espèces, et estimer ainsi quelles espèces sont les plus proches.
Les séquences de collagène du Brachylophosaurus canadensis et du Gallus gallus (poulet) ont été alignées ci-dessous : la présence d’un * signifie que les acides aminés sont identiques à cette position.
Combien de différences ?

La démarche est un peu fastidieuse mais surtout, elle n’est pas tout-à-fait finie : certains changements de séquence ont plus d’impact biologiquement que d’autres. Un peu comme dans un texte : certaines fautes d’orthographes modifieront le sens, d’autres pas !
C’est la raison pour laquelle les programmes bioinformatiques qui comparent les séquences utilisent des ‘matrices de substitution’.
Une valeur est attribuée à chaque paire d’acides aminés (par exemple : A-A: 4; G-G: 6; A-R: – 1). L’addition de ces valeurs permet de calculer le score de l’alignement.
Pour les experts: un exemple de matrice de substitution
Les matrices de substitution permettent de calculer le score d’un alignement en attribuant une valeur à chaque ‘paire’ d’acides aminés. Il existe différentes matrices de substitution qui ont été établies en étudiant l’évolution de plusieurs familles de protéines.
De façon générale, la valeur de substitution est basée sur le degré de similitude des deux acides aminés. S’ils sont très similaires (en termes de forme et de propriétés physicochimiques), la paire d’acides aminés est probable et donc recevra un score positif. Si les acides aminés sont très différents, la paire est improbable et recevra donc un score négatif. Un score de 0 correspond à une paire qu’on pourrait observer par chance.
Voici, par exemple, la matrice de substitution appelée BLOSUM62 :

Classification des dinosaures et des oiseaux : un exemple
L’arbre ci-dessous a été calculé en comparant les séquences de plusieurs protéines collagène provenant de plus de 30 espèces différentes. Il a été validé par des programmes statistiques complexes.
Image librement adaptée de Expansion for the Brachylophosaurus canadensis Collagen I Sequence and Additional Evidence of the Preservation of Cretaceous Protein (2017)
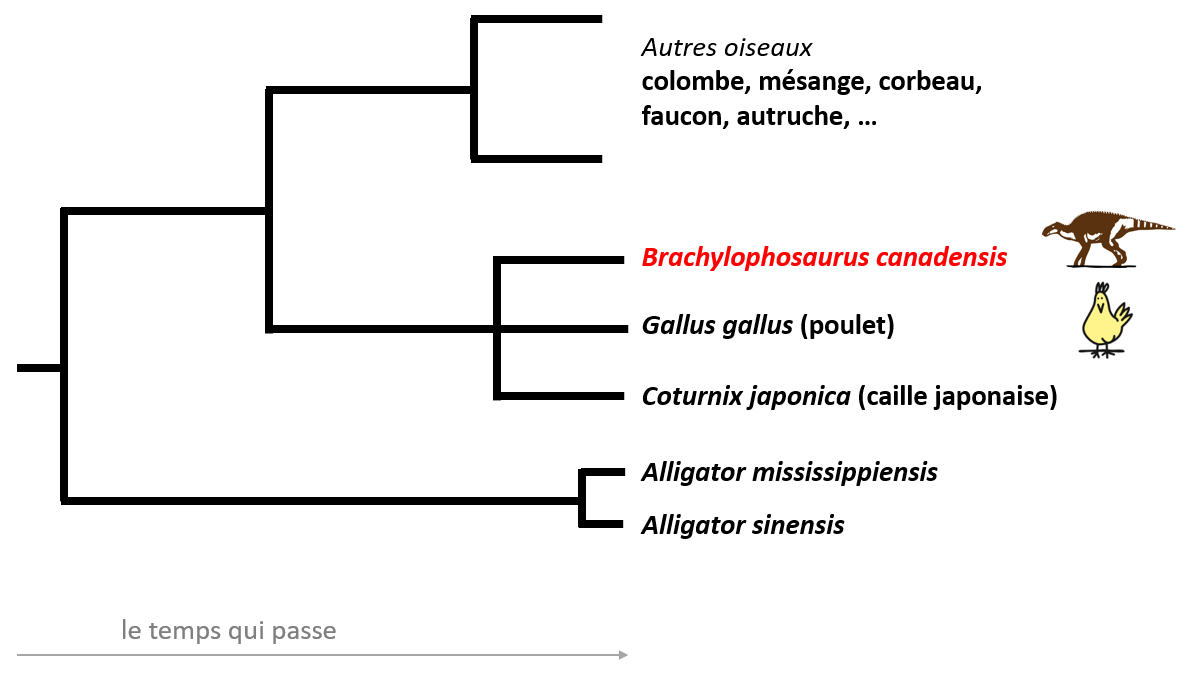
Ces analyses ont permis d’établir que les dinosaures sont apparentés aux oiseaux.
Ces résultats confirment les prédictions faites à partir des comparaisons anatomiques des squelettes. Il s’agit toutefois de la première preuve moléculaire (génétique) du lien évolutif qui existe entre les oiseaux modernes et les dinosaures.
Les dinosaures n’ont donc pas tous disparu : un petit groupe a survécu et parmi eux, les dinosaures ‘aviaires’, les ancêtres des oiseaux.
Source: Est-ce que les poules descendent vraiment des dinosaures ? (2014)
Un autre arbre plus détaillé pour comprendre les liens de parenté entre les oiseaux et les dinosaures (Wikimedia).
Le mystère de la plume
En 1861, quelques mois après la parution de l’ouvrage de Charles Darwin L’Origine des espèces, une plume fossile d’un peu moins de 7 cm et vieille de 150 millions d’années, a été découverte dans des carrières à Solnhofen, en Allemagne. L’identification du propriétaire de la plume a longtemps fait l’objet de controverses. Selon les dernières études, elle aurait appartenu à un Archaeopteryx lithographica, un dinosaure à plumes.
Source: Detection of lost calamus challenges identity of isolated Archaeopteryx feather (2019)
Nous savons aujourd’hui qu’Archaeopteryx lithographica est plus apparenté aux oiseaux qu’aux autres dinosaures. La classification des dinosaures et des oiseaux reste toutefois encore controversée !
Il y a 150 millions d’années, Archaeopteryx lithographica appartenait à une espèce parmi d’autres, espèce sans aucune destinée programmée !
Un petit résumé:
Archaeopteryx lithographica. Source: Wikipedia



