Dauer der Aktivität: 30 Minuten
Altersempfehlung: ab 15 Jahren
Comparative genomicsQuest for orthologs
Geschenkte Gene
– Jetzt sind Sie dran
Sushi, Fläschchen und vegetarisches Menü

Hintergrund
Der horizontale Gentransfer („Horizontal Gene Transfer“, HGT) spielt eine wichtige Rolle bei der Evolution von Organismen.
Was ist das?
Beim horizontalen Gentransfer baut ein Organismus DNA von einem anderen, nicht verwandten Organismus in seine eigene DNA ein.
Warum ist das wichtig?
So erwerben beispielsweise viele Bakterien durch HGT Antibiotikaresistenzgene. Einige Insekten stehlen Gene von ihrer Lieblingspflanze, um die Verteidigungsmechanismen der Pflanze anzugreifen. Sogar Viren können ihre Gene übertragen und haben dadurch die Evolution der Säugetiere geprägt – etwa 8 % des menschlichen Genoms sind viralen Ursprungs (siehe weiter unten).
Dazu kommt, dass horizontaler Gentransfer möglicherweise eine Schlüsselrolle gespielt hat bei der Entstehung von Sex. ( Zurück zu „Geschenkte Gene – Geschichte“)
Ein paar Beispiele: Horizontal Gene Transfer Happens More Often Than Anyone Thought (2022) / Slideshow: Examples of Eukaryotic Horizontal Gene Transfer (2022)
Japaner und Sushi: eine Geschichte über Bakterien
Horizontaler Transfer: Meeresbakterien – Landbakterien
Japaner essen durchschnittlich 14 Gramm Algen pro Tag, darunter auch die Rotalge, die manche Sushis umhüllt.
Diese Algen enthalten komplexe Zucker, die nur von Japanern verdaut werden können: warum?
In unserem Darm befinden sich etwa 10 Milliarden Bakterien, die sogenannte Darmflora oder intestinale Mikrobiota. Diese Bakterien unterstützen die Verdauung von Nahrungsbestandteilen, insbesondere von komplexen Zuckern, die eine wichtige Energiequelle darstellen.
Die Japaner sind die einzigen Menschen, die ein Darmbakterium namens B. plebeius besitzen. Dieses Bakterium hat jedoch mehrere Gene von Z. galactanivorans, einem Meeresbakterium, das auf Rotalgen vorkommt, „gestohlen“. Und diese Gene ermöglichen es B. plebeius, die komplexen Zucker zu verdauen, die in den für Sushirollen verwendete Rotalgen vorkommen.
Ergebnis: Japaner sind die einzigen, die Sushi richtig verdauen!
Die Darmflora der Japaner hat sich also an die in Sushi gefundenen komplexen Zucker angepasst und ist in der Lage, diese zu verdauen, dank (1) des Vorhandenseins des Bakteriums B. plebeius und (2) des horizontalen Gentransfers zwischen dem marinen Bakterium (Z. galactanivorans) und dem terrestrischen Bakterium (B. plebeius) (und der daraus resultierenden Kompetenz, komplexe Zucker abzubauen).
Jetzt sind Sie dran
Dies ist die Aminosäuresequenz des Proteins, das durch das PorB-Gen von B. plebeius codiert wird. Dieses Protein ermöglicht es den Japanern, die komplexen Zucker zu verdauen, die in den Sushi-Algen zu finden sind.
>sp|B5CY92|P.plebeius MRKTVLYLSAASLFLSSYTLKNDKEYSLAEEHIKNLPEAPEGYKWVVNEDYTDEFNGKRL NAAKWHAKSPYWTNGRPPATFKAENVSVKKGCLRIINTVLSPTEGLDGKPGDKYRLAGGA VASVKNQAHYGYYETRMKASLTTMSSTFWLSNRPVMKEIMKGGKKIKTWSSQELDIIETM GIIRSVNPDNPWNKTWNMQMNSNTHYWYQEQGGKRTDNTAKRSDVVSYMTDPSAEDFHTY GCWWVDANTVKFYYDGKYMYTIKPTTKYTDTPFDRPMFIHIVTETYDWEKQVPTAEDLKD KDKSTTYYDWVRAYKLVPIEE
Frage: Welchem Protein aus welcher anderen Spezies ähnelt es am meisten?
Um diese Frage zu beantworten, muss man eine „Ähnlichkeitssuche“ mithilfe eines Bioinformatik-Werkzeugs namens BLAST durchführen.
BLAST ermöglicht es, die Sequenz eines Proteins mit allen Sequenzen in einer Datenbank zu vergleichen und, falls vorhanden, die ähnlichsten Sequenzen in Sekundenschnelle zu finden. So kann man herausfinden, ob ein Protein in einer bestimmten Spezies vorkommt.
Anleitung:
- Kopieren sie die Sequenz und fügen Sie sie in das BLAST-Formular von UniProt ein: https://www.uniprot.org/blast
- Wählen Sie UniProtKB Swiss-Prot als „Target database“.
- Klicken Sie auf „Run Blast“.
Antwort
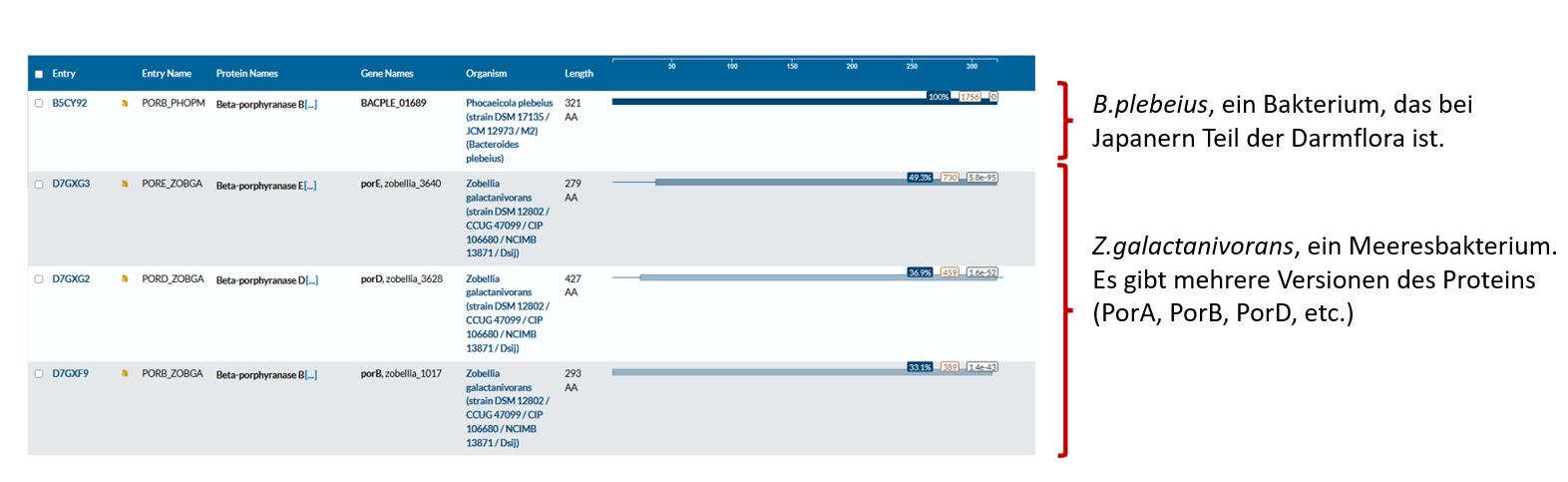
Das PorB-Protein von B. plebeius ist dem PorE-Protein von Z. galactanivorans am ähnlichsten, einem Meeresbakterium, das auf Rotalgen vorkommt.
Die Ähnlichkeit zwischen diesen Proteinen bestätigt, dass das Gen, das für dieses Protein kodiert, ursprünglich vom Meeresbakterium Z. galactanivorans stammt und auf das Landbakterium B. plebeius übertragen worden ist, ein Bakterium, das bei Japanern Teil der Darmflora ist.
Viren und Säugetiere: eine Geschichte über Verschmelzung
Horizontaler Transfer: Virus – Eukaryoten
Im Laufe der Evolution waren Wirbeltiere mehrfach Infektionswellen durch sogenannte Retroviren ausgesetzt. Diese Viren sind in der Lage, sich in die DNA des Wirts zu integrieren.
Einige Retroviren haben sich in die DNA der Keimzellen integriert und wurden so auf die folgenden Generationen übertragen.
Diese DNA-Sequenzen viralen Ursprungs werden als „endogen“ betrachtet und aus diesem Grund als ERV (für „Endogenous RetroVirus“) bezeichnet. Die DNA dieser integrierten Retroviren macht heute zwischen 8% und 10% des Genoms von Mensch und Maus aus!
Die DNA dieser Retroviren enthält Gene, die für Proteine kodieren. Diese retroviralen Gene sind also über Millionen von Jahren seit ihrer Integration funktionstüchtig geblieben.
Für die Experten : Der Mensch besitzt etwa 20.000 Gene. Hier ist eine Liste der etwa 80 menschlichen retroviralen Gene in UniProtKB.
Syncytine
Zu den menschlichen Proteinen, die von Sequenzen viralen Ursprungs produziert werden, gehören auch die Syncytine.
Diese Proteine haben fusogene Eigenschaften und spielen eine wichtige Rolle bei der Fusion bestimmter Zellen im Embryo, was für die Entwicklung der Plazenta unerlässlich ist.
Was ist mit dem Fläschchen?
Die Plazenta und die Laktation sind zwei wesentliche Bestandteile der Entwicklung von Säugetieren, und ihre Evolution ist eng miteinander verbunden.
Die Evolution der Plazenta ermöglichte die Entwicklung von Neugeborenen, die bei der Geburt weiter entwickelt waren. Die Evolution der Laktation ermöglichte es, diesen Neugeborenen eine geeignete Nahrungsquelle zur Verfügung zu stellen.
Ergebnis: Ohne Viren und horizontalen Gentransfer hätte es auf der Erde vielleicht keine Säugetiere gegeben!
Jetzt sind Sie dran
Hier ist die Aminosäuresequenz des Proteins Syncytin beim Menschen:
>sp|Q9UQF0|Syncytin-1 OS=Homo sapiens MALPYHIFLFTVLLPSFTLTAPPPCRCMTSSSPYQEFLWRMQRPGNIDAPSYRSLSKGTP TFTAHTHMPRNCYHSATLCMHANTHYWTGKMINPSCPGGLGVTVCWTYFTQTGMSDGGGV QDQAREKHVKEVISQLTRVHGTSSPYKGLDLSKLHETLRTHTRLVSLFNTTLTGLHEVSA QNPTNCWICLPLNFRPYVSIPVPEQWNNFSTEINTTSVLVGPLVSNLEITHTSNLTCVKF SNTTYTTNSQCIRWVTPPTQIVCLPSGIFFVCGTSAYRCLNGSSESMCFLSFLVPPMTIY TEQDLYSYVISKPRNKRVPILPFVIGAGVLGALGTGIGGITTSTQFYYKLSQELNGDMER VADSLVTLQDQLNSLAAVVLQNRRALDLLTAERGGTCLFLGEECCYYVNQSGIVTEKVKE IRDRIQRRAEELRNTGPWGLLSQWMPWILPFLGPLAAIILLLLFGPCIFNLLVNFVSSRI EAVKLQMEPKMQSKTKIYRRPLDRPASPRSDVNDIKGTPPEEISAAQPLLRPNSAGSS
Frage: In welchen Arten findet man ein Protein, das dem menschlichen Syncytin ähnlich ist?
Um diese Frage zu beantworten, müssen wir eine „Ähnlichkeitssuche“ mithilfe des BLAST-Tools durchführen.
Anleitung:
- Kopieren Sie die obige Sequenz und fügen Sie sie in das BLAST-Formular von UniProt ein: https://www.uniprot.org/blast
- Wählen Sie UniProtKB Swiss-Prot als „Target database“
- Klicken Sie auf „Run Blast“
Antwort
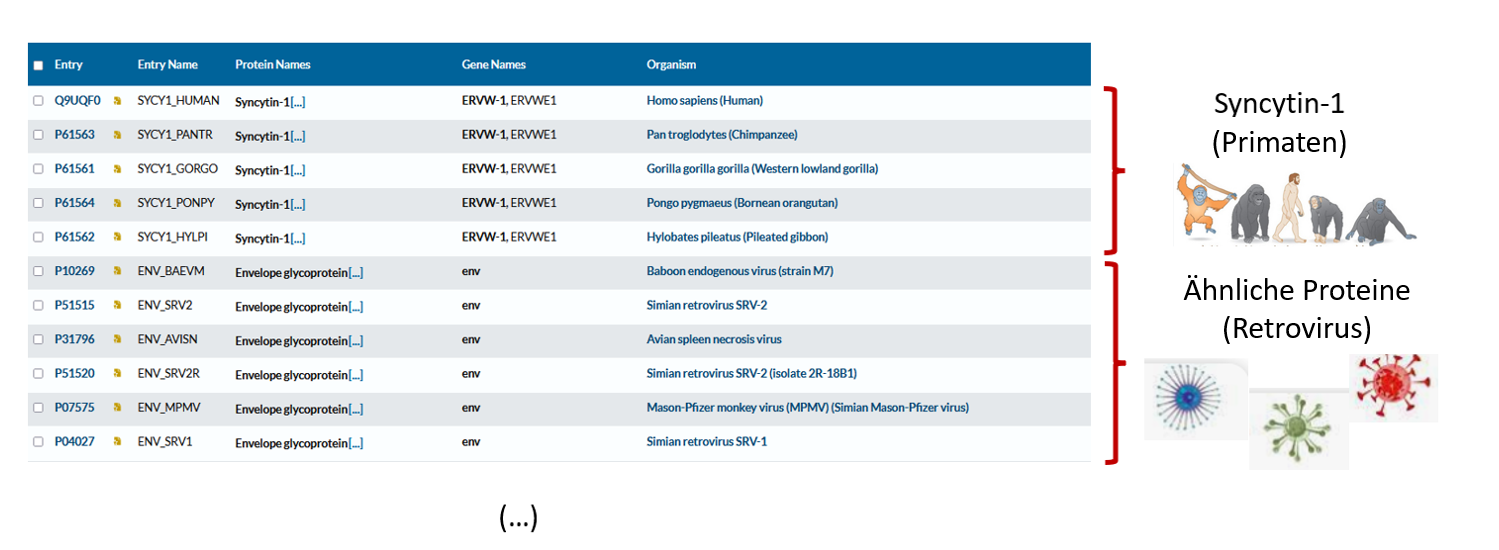
Die ähnlichsten Syncytine werden bei Primaten und bei Retroviren gefunden.
Ein Vorfahre der Primaten, der vor 45 bis 70 Millionen Jahren gelebt hat, wurde mit einem Retrovirus infiziert und erwarb dadurch die Gene, die für Syncytine kodieren.
Syncytine kommen bei allen Säugetieren vor: die Aufnahme der Syncytin-Gene erfolgte unabhängig voneinander durch verschiedene Retroviren in den verschiedenen Säugetierlinien vor etwa 10 und 85 Millionen Jahren. Die Syncytine anderer Säugetiere unterscheiden sich daher ein wenig und erscheinen im BLAST-Ergebnis „weiter hinten“.
Weitere Informationen
Endogenous Retroviruses in Trophoblast Differentiation and Placental Development (2010)
Pflanzen und Insekten: eine Geschichte der Gegenwehr
Horizontaler Transfer: Pflanze – Insekt
Insekten sind hauptsächlich Pflanzenfresser. Eine kleine weisse Fliege, B. tabaci, befällt über 600 Pflanzenarten und verursacht weltweit große Ernteausfälle.
Um sich gegen Insektenschädlinge zu wehren, stellen Pflanzen Insektizide her, insbesondere Moleküle, die als Phenylglykoside bezeichnet werden. Diese Moleküle beeinflussen das Wachstum, die Entwicklung und das Verhalten von pflanzenfressenden Insekten.
In hohen Dosen sind diese Moleküle aber auch für die Pflanze selbst giftig. Um sich zu schützen, besitzt die Pflanze, die diese Moleküle produziert, ein Protein, das die phenolischen Glykoside verändert. Dadurch kann die Pflanze diese Moleküle in einer ungiftigen Form speichern.
Ein ähnliches Protein (BtPMaT1-Gen) wurde bei der Weissen Fliege B. tabaci gefunden. Das Vorhandensein des BtPMaT1-Gens ermöglicht es dem Insekt, phenolische Glukoside in Pflanzen zu neutralisieren und so dem Insektizid der Pflanze zu widerstehen!
Das BtPMaT1-Protein aus B. tabaci hat kein Homolog in anderen Insekten, weist jedoch große Ähnlichkeiten mit anderen Proteinen pflanzlichen Ursprungs auf.
Es gibt nur eine Schlussfolgerung: das BtPMaT1-Gen wurde von der Weissen Fliege wahrscheinlich vor etwa 86 Millionen Jahren „horizontal“ aus Pflanzen erworben. Es wurde wahrscheinlich durch Virusinfektionen von den Pflanzen auf die Weisse Fliege übertragen, ein natürlicher Prozess, der beim horizontalen Gentransfer eine wichtige Rolle spielt.
Ergebnis: horizontaler Gentransfer ermöglichte es einigen Insekten, ihren vegetarischen Speiseplan zu variieren.
Jetzt sind Sie dran
Dies ist die Aminosäuresequenz des Proteins der Weißen Fliege. Dieses Protein sorgt dafür, dass die Fliege gegen das Insektizid in den Pflanzen resistent ist.
>sp|P0DUQ3|Bemisia tabaci MSISSSVAVLNVVQVSPPTAPVNNAFQDRISLTHFDLLALRAPPNQRLFFYETHLPISAF AETVIPKLRDSLSLTLQNFRPLAGTLIWSLHSDEPYIRIKDDDSVPLTIAETDADPQKLF DDPFQQETDLQQLLPPLRVSETEASLLALQITLFPSGDICLGITFHHAAQDGASLALFLK SWAHICRHGDDPPLPQNLIPIFDRDFIDDPKNIKQLFLDHLLTPLTPGGPRNRSVKPMEK PFNDRMHGSFRLTVDDIENLRRRITSLQVQNTSQEPPVRMSTVVVTCAYVLTCFVKAGLT KKHVRFILPADLRKRLQPPVPDNYYGNCVFGCTVDMSSDDLAGQDGLVVAAKTISSVVSE LDANDHRTFFENFLLNNTISQEETKVGVGGSIYFSLDEKDFGWGGPKHLKNVPPWPNHIY LAERRDGDKGVDFCLMLAKQEMAEFESKFLDDLKLLEKRSC
Frage: Welchem Protein welcher Art ähnelt es am meisten?
Um diese Frage zu beantworten, müssen wir eine „Ähnlichkeitssuche“ mithilfe des BLAST-Tools durchführen.
Anleitung:
- Kopieren Sie die Sequenz und fügen Sie sie in das BLAST-Formular von UniProt ein: https://www.uniprot.org/blast
- Wählen Sie UniProtKB Swiss-Prot als „Target database“.
- Klicken Sie auf „Run Blast“.
Antwort

Das BtPMaT1-Protein der Weissen Fliege B.tabaci sieht sich selbst am ähnlichsten…und Pflanzenproteinen!
Weitere Informationen
Whitefly hijacks a plant detoxification gene that neutralizes plant toxins (2021)
